DNA甲基化(5mC)和RNA甲基化(m6A)广泛存在于动植物,真菌以及原核生物中,其在多种生命进程中扮演着重要的角色1,2。调控5mC/m6A水平的修饰酶包括甲基转移酶DNMTs、METTL3/METTL14及去甲基化酶TETs、FTO、ALKBH5等。诸多研究表明,这些修饰酶与多种疾病发生发展密切相关,是重要的潜在疾病治疗靶点,具有极大的临床价值3-10。虽然如此,目前该领域只有靶向DNMT的两个核苷类药物上市,相关化合物发现研究进展缓慢。由于甲基是一种仅有15Da的非极性基团,难以被传统的荧光、OD等方法检测。因此,目前5mC和m6A甲基化酶的活性检测主要通过放射性同位素标记法进行11,12,而5mC和m6A去甲基化酶则几乎全都使用高效液相色谱定量法13,14。然而这些方法都是非均相的,且操作繁琐,费时费力,价格昂贵,这些问题都限制了其应用于大规模自动化的化合物高通量筛选。缺乏高效、高通量的酶活评价方法是限制5mC和m6A修饰酶药物开发的关键障碍之一。
针对此问题,基于课题组与合作课题组在核酸修饰研究的前期工作(Nature. 2015 Nov 5;527(7576):118-22.;J Am Chem Soc. 2012 Oct 31;134(43):17963-71;Nucleic Acids Res. 2015 Jan;43(1):373-84;Chem Sci. 2019 Jun 27;10(31):7407-7417;Phys Chem Chem Phys. 2016 Feb 14;18(6):4728-38;ACS Chem Biol. 2018 Mar 16;13(3):772-781;J Med Chem. 2014 Nov 13;57(21):9028-41),中科院上海药物所罗成课题组陈示洁副研究员和萧森豪博士开发了一种简单、高效且实现高通量的5mC/m6A核酸修饰酶的活性检测方法,该研究工作已于2021年10月29日在线发表于Nucleic Acids Research(High-Throughput-Methyl-Reading (HTMR) Assay: a solution based on Nucleotide Methyl-Binding Proteins enables largescale screening for DNA/RNA Methyltransferases and Demethylases)。
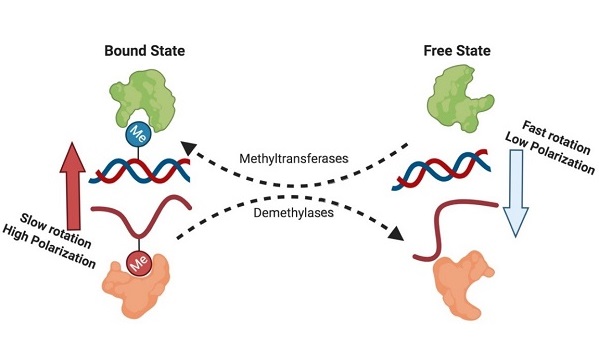
图 1. 5mC/m6A核酸修饰酶的活性检测原理图
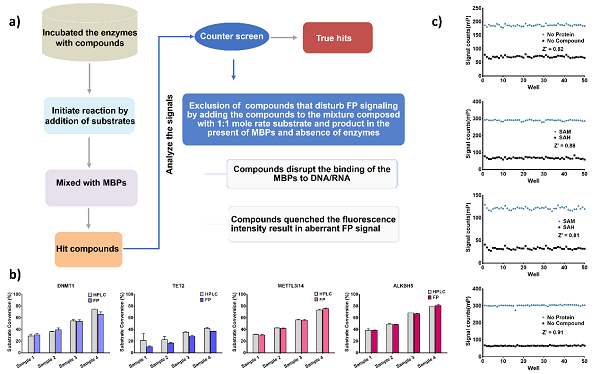
在该研究中,研究人员利用在细胞内本身存在的一类“化学物质”(甲基化阅读器蛋白:UHRF1,MBPs,YTH家族蛋白等)作为甲基的识别蛋白,它们可以对不同甲基化状态的DNA/RNA产生不同的响应(蛋白-底物核酸结合的热力学过程)。随后,研究人员将这一热力学过程与荧光偏振实验相偶联,建立了一种均相的-“高通量甲基阅读”(High-Throughput-Methyl-Reading, HTMR)分析方法:首先将底物进行荧光标记,经过酶催化后加入甲基结合蛋白MBD1和YTHDF1。由于底物和产物修饰状态不同,通过对结合蛋白大小、浓度及结合条件进行摸索及优化,使得结合蛋白对底物和产物的亲和力产生显著差异,其形成的底物/产物探针-结合蛋白复合物产生荧光偏振信号值,可以通过检测得到的荧光偏振信号值,表征体系中酶的活性。该方法原理经过热力学计算验证可行。通过将5mC和m6A的甲基化状态与特定甲基结合蛋白的荧光偏振结合实验产生的荧光信号相偶联,科研人员实现了对这些核酸甲基化修饰酶如DNMT1,TET2,METTL3-METTL14以及ALKBH5的活性检测,并将其应用在这些蛋白的高通量筛选上。实验结果表明,HTMR方法与平行的HPLC定量实验的检测误差在3%以内,证明了该方法的准确性和可靠性。同时,该方法可以对阳性抑制剂进行准确的评价,且非常适合于开展底物竞争实验以探究化合物的分子作用机制。该方法具有很高的Z-factor,表现出很好的稳定性。此外,该方法还可以帮助排除一些与核酸结合的假阳性化合物。
图 2. a) HTMR方法的实验操作与质量控制流程;b) 平行的甲基化/去甲基化HPLC定量实验;c) HTMR方法的稳定性评估实验;
综上,本研究提供了一种简单、快速、均相、低成本、高灵敏度、高通量的5mC/m6A修饰酶的活性检测方法-HTMR。该方法不需要经历固定,洗涤,转移,酶处理等繁琐的处理过程,仅需要在酶反应完成后,将检测试剂-即特定浓度的甲基结合蛋白引入到反应体系中,即可产生最终的信号并完成检测,非常适合应用于5mC/m6A核酸修饰酶的化合物高通量筛选研究。该项研究将为靶向DNA/RNA甲基化的小分子调控剂的发现及活性评价提供强有力的工具。
本课题在上海药物所罗成研究员的协助下完成,罗成课题组的陈示洁副研究员为该论文的通讯作者,上海药物所和上海科技大学联合培养博士萧森豪为该论文第一作者。该研究还得到华中农业大学殷平教授、复旦大学徐彦辉教授、上海交通大学张良教授、上海药物研究所陈凯先院士、蒋华良院士、周虎研究员、杨财广研究员、张乃霞研究员的支持。研究得到国家自然科学基金生物大分子动态修饰重大科学计划、杰出青年基金、重点国际(地区)合作研究项目、青年基金和上海市科委等项目的资助。
全文链接:
参考文献:
1 Jones, P. A. Functions of DNA methylation: islands, start sites, gene bodies and beyond. Nature Reviews Genetics 13, 484-492, doi:10.1038/nrg3230 (2012).
2 Zaccara, S., Ries, R. J. & Jaffrey, S. R. Reading, writing and erasing mRNA methylation. Nat Rev Mol Cell Bio 20, 608-624, doi:10.1038/s41580-019-0168-5 (2019).
3 Huang, Y. et al. Small-Molecule Targeting of Oncogenic FTO Demethylase in Acute Myeloid Leukemia. Cancer Cell 35, 677-691 e610, doi:10.1016/j.ccell.2019.03.006 (2019).
4 Li, Z. et al. FTO Plays an Oncogenic Role in Acute Myeloid Leukemia as a N(6)-Methyladenosine RNA Demethylase. Cancer Cell 31, 127-141, doi:10.1016/j.ccell.2016.11.017 (2017).
5 Barbieri, I. et al. Promoter-bound METTL3 maintains myeloid leukaemia by m(6)A-dependent translation control. Nature 552, 126-131, doi:10.1038/nature24678 (2017).
6 Huang, Y. et al. Meclofenamic acid selectively inhibits FTO demethylation of m6A over ALKBH5. Nucleic Acids Res 43, 373-384, doi:10.1093/nar/gku1276 (2015).
7 Lyko, F. The DNA methyltransferase family: a versatile toolkit for epigenetic regulation. Nat Rev Genet 19, 81-92, doi:10.1038/nrg.2017.80 (2018).
8 Constantinides, P. G., Jones, P. A. & Gevers, W. Functional striated muscle cells from non-myoblast precursors following 5-azacytidine treatment. Nature 267, 364-366, doi:10.1038/267364a0 (1977).
9 Cimmino, L. et al. Restoration of TET2 Function Blocks Aberrant Self-Renewal and Leukemia Progression. Cell 170, 1079-1095 e1020, doi:10.1016/j.cell.2017.07.032 (2017).
10 Agathocleous, M. et al. Ascorbate regulates haematopoietic stem cell function and leukaemogenesis. Nature 549, 476-481, doi:10.1038/nature23876 (2017).
11 Gros, C. et al. Development of a universal radioactive DNA methyltransferase inhibition test for high-throughput screening and mechanistic studies. Nucleic Acids Res 41, e185, doi:10.1093/nar/gkt753 (2013).
12 Li, F. et al. A Radioactivity-Based Assay for Screening Human m6A-RNA Methyltransferase, METTL3-METTL14 Complex, and Demethylase ALKBH5. J Biomol Screen 21, 290-297, doi:10.1177/1087057115623264 (2016).
13 Hu, L. et al. Crystal structure of TET2-DNA complex: insight into TET-mediated 5mC oxidation. Cell 155, 1545-1555, doi:10.1016/j.cell.2013.11.020 (2013).
14 Gerken, T. et al. The obesity-associated FTO gene encodes a 2-oxoglutarate-dependent nucleic acid demethylase. Science 318, 1469-1472, doi:10.1126/science.1151710 (2007).